2018年7月28日,中南大学湘雅医院、温州医科大学、中科院北京生命科学研究院等单位合作在国际权威学术期刊Nucleic Acids Research (IF=11.561)在线发表了题为“Performance evaluation of pathogenicity-computation methods for missense variants”的最新研究成果。
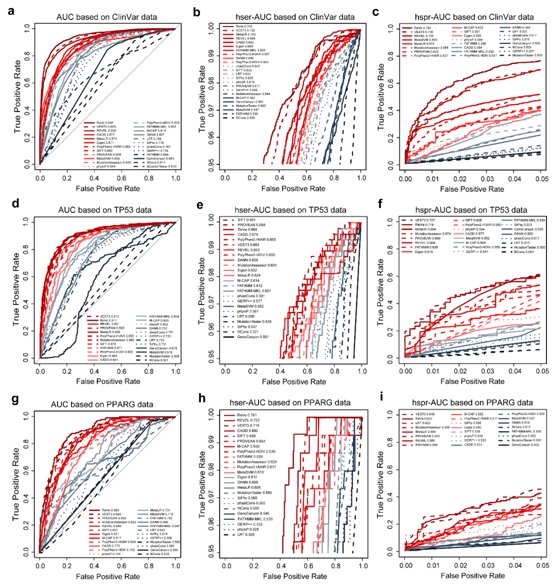
随着高通量测序技术在基因检测、遗传咨询以及医学遗传学研究中得到了广泛应用,国际上多个团队开发了一系列针对错义突变的致病性预测软件。尽管这些软件在公开发表的时候都声称具有最好的性能,但不同软件在不同条件下通常会表现出不同的准确性和灵敏度,选择高可信度的致病性预测软件显得尤为重要。因此,我们研究团队站在第三方的角度,分别选取了四种不同的测试数据对23款软件的12项指标进行了系统评估。这四类测试数据包括:(1)ClinVar数据库中遗传疾病相关的生殖系突变;(2)TP53 和ICGC数据库中癌症相关的体细胞突变;(3)经过功能实验验证的突变;(4)孤独症相关的新发突变。
经过系统的比较分析,我们发现:(1)大部分软件在不同条件下表现出不同的性能,表明这些软件具有不同的适用条件。(2)不同的测试数据都证明这些软件预测致病突变的特异性明显低于敏感度,表明我们在数据分析中应当设置更严格的阈值来鉴别致病突变。(3)常用的软件,例如SIFT,PolyPhen2,MutationTaster表现出中等水平的准确性,大多数情况不推荐使用。(4)在所有四类测试数据中,REVEL和VEST3的多项指标最高,大多数情况下推荐用户使用。(5)我们还整合REVEL和VEST3这两款软件并开发出名为ReVe的新软件,综合评估中表现出最好的整体性能。我们已经计算出人类参考基因组所有错义突变的ReVe预测得分,供广大用户免费下载使用。总之,我们分析了各种条件下不同方法的性能,为最佳软件的选择提供了科学指导。
中南大学湘雅医院国家老年疾病临床医学研究中心的特聘教授李津臣博士与温州医科大学的硕士生赵婷婷为该研究论文的并列第一作者,李津臣博士与中科院北京生命科学研究院的孙中生教授为该论文的并列通讯作者。该研究获得国家重点研发计划和国家自然科学基金的支持。
发表回复
要发表评论,您必须先登录。